Roche uPath PD-L1 image analysis for NSCLC IVD Algorithm Användarmanual
- Typ
- Användarmanual

uPath PD-L1 (SP263) bildanalys
för icke-småcellig lungcancer
algoritmguide


Innehållsförteckning
Inledning 1
Översikt och förklaring av algoritm 2
Avsedd användning 3
Produktens avsedda användning 3
Syftet med algoritmguiden 3
Klinisk signifikans 4
Analysprinciper 5
Begränsningar 6
Datasäkerhet 7
Arbetsflöde för att använda uPath PD-L1-bildanalys för
NSCLC-algoritmen 8
Patologarbetsflöde 9
PD-L1-bildanalys 12
PD-L1-utvärdering 15
Utvärdering av PD-L1-algoritmfärg 15
Färgningsegenskaper 15
Prestandaegenskaper 26
Metodjämförelse 26
Patologens reproducerbarhetsstudier 27
Reproducerbarhetsstudier för skanner 28
Felsökning 29
Referanser 33


Inledning
Roche uPath enterprise software (uPath enterprise software)
med uPath PD-L1 (SP263) bildanalys för NSCLC-algoritmen
(icke-småcellig lungcancer) (uPath PD-L1 (SP263) bildanalys
för NSCLC-algoritmen) är ett programvarusystem utformat
för att stöda den kvantitativa bedömningen av proteinuttryck
i immunohistokemiskt (IHC) färgade histologiska sektioner
från formalinfixerade, paraffin-inbäddade (FFPE) normala och
neoplastiska vävnader.
uPath enterprise software är heltäckande programvara för digital
patologi som gör det möjligt för patologilaboratorier att inhämta,
hantera, visa, analysera, dela och rapportera om digitala bilder
av patologiprover. Med uPath enterprise software kan patologen
visa digitala bilder i olika förstoringar, lägga till kommentarer, göra
mätningar av vävnadssektioner, utföra bildanalys och generera
rapporter.
Obs! uPath PD-L1 (SP263) bildanalys för NSCLC-algoritmen är
en kompletterande datorassisterad metodik för att underlätta
inhämtning och mätning av bilder från objektglas för mikroskop
av vävnadsprover som IHC-färgats för förekomst av PD-L1-protein.
Giltigheten av bildanalysens poäng säkerställs genom att
patologen verifierar överenskommelse med lämpliga kontroller
som anges i metodbladet VENTANA PD-L1 (SP263) Rabbit
Monoclonal Primary Antibody (på www.ventana.com).
uPath PD-L1 (SP263) bildanalys för NSCLC (icke-småcellig lungcancer) algoritmguide 1

2 uPath PD-L1 (SP263) bildanalys för NSCLC (icke-småcellig lungcancer) algoritmguide
Översikt och förklaring av algoritm
För tillämpningar med bildanalys kan patologen använda
uPath enterprise software för att välja och skissera en eller flera
intressanta områden (ROI), och varje ROI kan ses med olika
förstoringar och sedan analyseras med uPath PD-L1 (SP263)
bildanalys för NSCLC-algoritmen. En summa av det totala antalet
mål-TC genereras och tumörceller (TC) stratifieras efter huruvida
de är färgade eller ofärgade. uPath PD-L1 (SP263) bildanalys
för NSCLC-algoritmen delar de färgade TC med det totala
antalet TC (färgade och ofärgade) för att generera PD-L1 TC-
positivitetspoäng på en skala från 0–100%. uPath PD-L1 (SP263)
bildanalys för NSCLC-algoritmen kan generera en poäng för en
viss ROI, eller en sammanlagd poäng för alla utvalda ROI på den
bilden. Även om algoritmen upptäcker icke-tumörceller som en
del av den totala analysen, visar överläggningen och utmatningen
endast TC som används vid beräkningen av TC-positivitetspoäng.
Patologen kan acceptera poängen som tillhandahålls av uPath
PD-L1 (SP263) bildanalys för NSCLC-algoritmen, eller åsidosätta
poängen med en annan poäng. Patologen måste noggrant
granska vad algoritmen har markerat som positiva och negativa
TC och bekräfta att algoritmen är korrekt eller ge en manuell
åsidosättning. uPath PD-L1 (SP263) bildanalys för NSCLC-
algoritmen gör inte oberoende tolkningar av data och ska därför
endast användas av en kvalificerad patolog i samband med
histologisk undersökning, relevant klinisk information och korrekt
kontroll. Det är utformat och indikerat som ett hjälpmedel för
patologen för bedömning av PD-L1-uttryck på 50%.

Avsedd användning
Syftet med algoritmguiden
Denna guide till uPath PD-L1 (SP263) bildanalys för NSCLC
algoritmguide (algoritmguide) är avsedd att:
• Ge bakgrundsinformation om den avsedda användningen
av uPath PD-L1 (SP263) bildanalys för NSCLC-algoritmen,
testprinciper och begränsningar.
• Definiera nödvändigt material, IT, datasäkerhet och
nätverkskrav.
• Visa stegvis vägbeskrivning för att köra uPath PD-L1 (SP263)
bildanalys för NSCLC-algoritm.
• Tillhandahåll fotografiska bilder som illustrerar hur uPath PD-
L1 (SP263) bildanalys för NSCLC-algoritmen ska användas.
• Ge patologer ett verktyg för att underlätta användningen av
uPath PD-L1 (SP263) bildanalys för NSCLC-algoritmen på
FFPE NSCLC-sektioner färgade med VENTANA PD-L1 (SP263)
Assay.
• Tillhandahåll exempelbilder på utmanande fall för att ge
vägledning om hur du använder uPath PD-L1 (SP263)
bildanalys för NSCLC-algoritmen i deras utvärdering.
• Visa prestandaegenskaper för uPath PD-L1 (SP263) bildanalys
för NSCLC-algoritmen.
Produktens avsedda användning
uPath PD-L1 (SP263) bildanalys för NSCLC-algoritmen är avsedd
att användas som ett hjälpmedel för patologen vid detektering
och semikvantitativ mätning av PD-L1-protein i formalin-fixerad,
paraffin-inbäddad NSCLC-vävnad. När den används med
VENTANA PD-L1 (SP263) Assay, är den indikerad för användning
som ett hjälpmedel för att identifiera patienter med NSCLC för
behandling med terapier med ≥ 50% PD-L1 TC-positivitetsgräns i
enlighet med den godkända terapeutiska produktmärkningen.
Obs! uPath PD-L1 (SP263) bildanalys för NSCLC är en
kompletterande datorassisterad metodik för att underlätta
inhämtning och mätning av bilder från objektglas för mikroskop
av vävnadsprover som IHC-färgats för förekomst av PD-L1-
protein. Giltigheten av bildanalysens poäng säkerställs genom
att patologen verifierar överenskommelse och använder lämpliga
kontroller som anges i metodbladet VENTANA PD-L1 (SP263)
Assay (art. nr. 741-4905).
Denna algoritm är avsedd för in vitro-diagnostisk användning
(IVD).
uPath PD-L1 (SP263) bildanalys för NSCLC (icke-småcellig lungcancer) algoritmguide 3

Klinisk signifikans
Lungcancer har varit den vanligaste cancerformen i världen
under flera decennier, och är fortfarande den ledande orsaken
till cancerdöd över hela världen. Det beräknas stå för 12,9% av
alla nya cancerfall och ligger bakom nästan 1,76miljoner dödsfall
årligen över hela världen, eller ungefär var femte cancerrelaterade
dödsfall. NSCLC är den vanligaste typen av lungcancer som
står för cirka 85% av alla fall av lungcancer. Flertalet patienter
med NSCLC uppvisar inoperabel, lokalt framskriden sjukdom
(Stadium IIIB) eller metastaserande sjukdom (Stadium IV), vilka
för närvarande båda saknar botande behandlingsalternativ. Den
5-åriga relativa överlevnadsgraden för NSCLC som diagnostiserats
i framskridna stadier är 4,7%.
Den programmerade death-ligand 1 (PD-L1) är ett
transmembranprotein uttryckt på aktiverade T-celler som
nedreglerar immunsvaret genom att binda till dess två hämmande
receptorer med programmerad death-1 (PD-1) och B7-1 (CD80).
Bindning av PD-L1 med PD-1 hämmar T-cellproliferation,
cytokinproduktion och cytolytisk aktivitet, vilket leder till
funktionell inaktivering eller utmattning av T-celler. PD-L1 binder
också med CD80 på antigenpresenterande celler och aktiverade
T-celler som medierar nedreglering av immunsvar, inklusive
hämning av T-cellaktivering och cytokinproduktion. PD-L1-
uttryck observeras i immunceller och TC. Avvikande uttryck av
PD-L1 på TC har rapporterats hindra anti-tumörimmunitet, vilket
resulterar i immundundation. Framkomsten av immunoterapier
som avbryter PD-L1/PD-1-vägen har visat sig förbättra
överlevnadshastigheten för patienter som diagnostiserats med
NSCLC. Prognos med behandling är emellertid beroende av nivån
på PD-L1-uttryck och kräver därför kvantifiering av PD-L1 med
immunohistokemisk färgning.
Immunohistokemi kan användas för att upptäcka specifika
antigener som finns i vävnadsprover, vilket gör det till ett
effektivt verktyg för patologer i deras diagnos och prognos för
karcinomer. VENTANA PD-L1 (SP 263) Assay är en monoklonal
kaninantikropp avsedd för laboratoriebruk för semikvantitativ
detektion av PD-L1-protein i sektioner av FFPE-normal och
neoplastisk vävnad. En fördel med histologiska vävnadspreparat
är att intakt vävnad tillåter morfologi att underlätta tolkningen av
PD-L1-positiviteten hos patientprovet. Alla histologiska tester
bör tolkas av en specialist i NSCLC-morfologi och/eller patologi.
Resultaten ska kompletteras med morfologiska studier, korrekt
kontroll och användas tillsammans med andra kliniska data och
laboratoriedata.
4 uPath PD-L1 (SP263) bildanalys för NSCLC (icke-småcellig lungcancer) algoritmguide

Analysprinciper
uPath enterprise software med uPath PD-L1 (SP263) bildanalys för
NSCLC-algoritmen använder bildanalystekniker för att erhålla en
TC-positivitetspoäng.
uPath PD-L1 (SP263) bildanalys för NSCLC-algoritmen använder
fördefinierade parametrar för att göra bilder av vävnad färgade
med VENTANA PD-L1 (SP263) Assay.
Steg involverade i bildanalys:
• Identifiering av vitt utrymme och automatisk uteslutning från
analys.
• Upptäckt av celler över hela bilden.
• Klassificering av celler som TC eller andra celltyper.
• Identifiering av färgade kontra ofärgade TC.
• Beräkning av TC-positivitetspoäng genom att dela antalet
färgade TC med det totala antalet TC enligt metodbladet för
VENTANA PD-L1 (SP263) Assay.
Så identifierar uPath PD-L1 (SP263) bildanalys för NSCLC-
algoritmen TC och hur så beräknas poängen:
• uPath PD-L1 (SP263) bildanalys för NSCLC-algoritm
identifierar TC med hjälp av färg, intensitet, storlek och
morfologiska funktioner.
• Identifierade TC klassificeras som färgade med detekterade
membran och förinställda trösklar som är i linje med
metodbladet för VENTANA PD-L1 (SP263) Assay.
• Procentandelen färgade TC beräknas genom att dela färgade
TC med det totala antal TC enligt metodbladet för VENTANA
PD-L1 (SP263) Assay.
Fastställande av vitt utrymme med uPath PD-L1 (SP263)
bildanalys för NSCLC-algoritmen:
• Algoritmen utesluter vitt utrymme automatiskt. Artefakter som
smuts, repor eller bläck kanske inte automatiskt utesluts.
• Användaren kan granska områdena som fastställs av
algoritmen för att vara vita utrymmen med ett falskt
färgöverlagring. Se ”PD-L1-bildanalys: Falsk färgöverlagring”.
uPath PD-L1 (SP263) bildanalys för NSCLC-algoritmens
kriterier för att generera utdata för TC-positivitetspoäng:
• uPath PD-L1 (SP263) bildanalys för NSCLC-algoritmen
rapporterar en poäng med en siffra som har en decimal,
t.ex. ”4,8%”.
• Vi rekommenderar mot att du rundar poängen upp eller ner. Till
exempel ska ”4,8%” inte rundas upp till 5%.
uPath PD-L1 (SP263) bildanalys för NSCLC (icke-småcellig lungcancer) algoritmguide 5

uPath PD-L1 (SP263) bildanalys för NSCLC algoritmen
är utformad för att fungera för VENTANA PD-L1 (SP263)
Assay. Testresultaten är endast så goda som kvaliteten och
noggrannheten på IHC-objektglaset som avbildas och den
efterföljande bilden som analyseras.
Patologen måste validera VENTANA PD-L1 (SP263) Assay-
färgning som utförs genom manuell mikroskopisk undersökning
av PD-L1-kontrollglasen för att verifiera att de förväntade
resultaten har uppnåtts innan bilder av objektglas ansluts till
uPath enterprise software för analys.
Använd tillverkarens rekommendationer för VENTANA
PD-L1 (SP263) Assay, inklusive alla positiva och negativa
kvalitetskontrollmaterial för varje färgningskörning. Om
kontrollglasen inte är acceptabla med manuell mikroskopisk
undersökning, ska vävnaden färgas igen med acceptabla resultat.
Patologen måste följa rekommendationerna för VENTANA PD-L1
(SP263) Assay-tolkning.
Se metodbladet för VENTANA PD-L1 (SP263) Assay
(art. nr. 1014258SV) och tolkguiden (art. nr. 1015317EN)
(finns på www.ventana.com).
uPath PD-L1 (SP263) bildanalys för NSCLC-algoritmen är
utformad för användning av en kvalificerad patolog i samband
med histologisk undersökning, relevant klinisk information och
korrekt kontroll. Den är inte utformad att vara ett fristående
verktyg och kräver kompetent mänskligt ingripande under hela
analysprocessen.
uPath PD-L1 (SP263) bildanalys för NSCLC-algoritmen kan
generera felaktiga poäng om de tagna bilderna har onormal
färgning (nukleär, cytoplasma, o.s.v.).
uPath PD-L1 (SP263) bildanalys för NSCLC-algoritmen avvisar
tumörkärnor som är långsträckta oavsett av cellens övergripande
form. Därför kan tumörer som innehåller ett stort antal celler med
avlånga kärnor behöva bedömas manuellt.
Begränsningar
uPath PD-L1 (SP263) bildanalys för NSCLC-algoritmen har
tränats, utvecklats och validerats på NSCLC-vävnadsprover.
uPath PD-L1 (SP263) bildanalys för NSCLC-algoritmen har
inte testats, eller så har inte dess säkerhet och effektivitet har
validerats, när den används med en persondator (PC) hemifrån.
uPath PD-L1 SP263-algoritmen för bildanalys är indikerad för
användning som ett hjälpmedel för att identifiera patienter
med NSCLC för behandling med terapier med 50% PD-L1
TC-positivitetsgräns i enlighet med den godkända terapeutiska
produktmärkningen.
uPath PD-L1 (SP263) bildanalys för NSCLC-algoritmen kan
felidentifiera celler på grund av närvaron av svag cytoplasmisk
färgning och/eller membranfärgning, stark immuncellfärgning
överlappande med tumörcellfärgning där det finns betydande
blandad inflammation, TC med cytoplasmisk rodnad och icke-
målfärgning. Detta kan leda till felidentifiering av TC som icke-TC
och av icke-TC som positiv TC.
Även om makrofager måste exkluderas från analysområdet är
det inte alltid möjligt att exkludera alla makrofager. Därför kan
poängen som genereras av uPath PD-L1 (SP263) bildanalys
för NSCLC-algoritmen påverkas av makrofagerna i ROI som
analyseras. Detta är avgörande när en patientpoäng är nära 50%.
Cytoplasmisk färgning är i allmänhet diffus med vissa fall med
en fin granulär kvalitet. Sällsynta fall har visat en perinukleär
prickliknande kroppsfärgning med variabel intensitet. Den
totala procentandelen av tumörmembranets signalintensiteter
uppskattas visuellt och används för att generera PD-L1-
uttrycksnivån. Cytoplasmisk färgning av tumörceller beaktas
för att fastställa PD-L1-uttryck. En isotypmatchad negativ
kontrollantikropp används för att utvärdera närvaron av bakgrund
i testprover och upprätta en baslinje för färgningsintensitet.
6 uPath PD-L1 (SP263) bildanalys för NSCLC (icke-småcellig lungcancer) algoritmguide

Datasäkerhet
Skadlig programvara eller icke-auktoriserad åtkomst till
instrumentet kan leda till förlust av data eller driftavbrott på
instrumentet.
Följande rekommendationer är viktiga för att undvika infektion
med skadlig programvara eller obehörig åtkomst och missbruk av
instrumentet:
• Installera inte och kör inte någon annan programvara på
instrumentet.
• Kontrollera att andra datorer och tjänster på nätverket
är korrekt säkrade och skyddade mot skadlig
programvara och icke-auktoriserad åtkomst. Till exempel
laboratorieinformationssystemet (LIS), delad arkivering, delad
säkerhetskopiering eller service.
• Kunderna är ansvariga för säkerheten i deras lokala nätverk,
särskilt för att skydda det mot skadlig programvara och
attacker. Detta skydd kan inkludera åtgärder som en
brandvägg, för att skilja enheten från okontrollerade nätverk.
Skyddet kan också inkludera åtgärder som garanterar att det
anslutna nätverket är fritt från skadlig kod.
• Begränsa fysisk åtkomst till instrumentet och all ansluten IT-
infrastruktur (datorer, kablar, nätverksutrustning osv.).
• Se till att instrumentets säkerhetskopior och arkivfiler
är skyddade från obehörig åtkomst och haveri. Denna
lista innefattar fjärrlagringsplats, webbplatser för
haveriåterställning och säker överföring av säkerhetskopior.
• Använd en brandvägg om möjligt, för att begränsa
nätverkstrafiken.
• USB-minnen kan användas för flera typer av säkerhetskopior
och återställningar. Felaktig hantering av ett USB-minne kan
leda till dataförlust eller fel på instrumentet.
• Använd endast USB-minnen som testas och installeras av din
lokala Roche-servicerepresentant.
• Endast en USB-enhet kan användas åt gången. Innan du sätter
i ett USB-minne ska du kontrollera att ingen annan USB-enhet
är insatt.
• Innan du tar bort ett USB-minne ska du klicka på knappen
Eject (Mata ut) i Windows.
• Standardinställningen för operativsystem (OS) som medföljer
servern bör inte ändras eftersom det påverkar de inställda OS-
konfigurationerna.
• Använd USB-flashenheten uteslutande på instrumentet för
att förhindra att ett virus infekterar uPath enterprise software.
Förvara inte andra data på detta USB-minne.
uPath PD-L1 (SP263) bildanalys för NSCLC (icke-småcellig lungcancer) algoritmguide 7

Arbetsflöde för att använda uPath PD-L1-bildanalys för NSCLC-algoritmen
Material som behövs
• uPath enterprise software
• uPath PD-L1 (SP263) bildanalys för NSCLC-algoritmen
• NSCLC-vävnadsglas som är färgade med VENTANA PD-L1 (SP263) Assay (med OptiView DAB IHC Detection Kit) på BenchMark
ULTRA-instrumentet
• VENTANA DP 200-ojbektglasskanner
Arbetsflöde
1. Ett NSCLC-vävnadsprov på ett glasobjekt färgas med VENTANA PD-L1 (SP263) Assay med användning av ett BenchMark ULTRA-
instrument.
2. Bildförvärv (hel bildskanning) utförs med VENTANA DP 200-bildskannern med 20x förstoring i ett z-plan.
3. När digitala bilder har inhämtats överförs dessa bilder från datorn som är associerad med VENTANA DP 200-bildskannern till
bildhanteringssystemet (IMS) på en centraliserad server.
4. Efter överföring till servern skapas ett fall i uPath enterprise software. Skapande av fall kan ske automatiskt genom kommunikation
med laboratorieinformationssystemet (LIS) med identifieringsinformation (d.v.s vävnadstyp och primär antikropp) som finns i
streckkodsetiketten på objektglaset eller matas in manuellt i uPath enterprise software (se uPath enterprise softwares handbok
(art. nr. 1018943SV)).
5. Om uPath PD-L1 (SP263) bildanalys för NSCLC-algoritmen är installerad (måste installeras på en separat server från uPath
enterprise software och IMS) och en 20x-bild inhämtas med lämplig färg och vävnadstyp, uPath enterprise software utlöser sedan
automatiskt helbildsanalys (WSA).
6. WSA analyserar automatiskt hela den skannade bilden.
7. När WSA är klar meddelas patologen i uPath enterprise software att ”analysis is complete” (analysen är klar). Patologen kan nu
välja specifika ROI:er att poängsätta. En patolog kan välja en enda ROI av valfri storlek eller flera ROI:er. Om flera ROI:er väljs
tillhandahålls en sammanlagd poäng samt en individuell poäng för varje ROI.
Färgning
• Vävnadsberedning och färgning ska följa rekommendationerna i metodbladet för VENTANA PD-L1 (SP263) Assay.
• Alla korrekta kontroller ska granskas och objektglasen ska återställas om färgningen inte uppfyller riktlinjerna i metodbladet för
VENTANA PD-L1 (SP263) Assay.
• uPath PD-L1 (SP263) bildanalys för NSCLC-algoritmen kräver användning av VENTANA PD-L1 (SP263) Assay, och allt ytterligare
material eller tillbehör som anges i metodbladet för VENTANA PD-L1 (SP263) Assay, för att färga vävnader före analys. VENTANA
PD-L1 (SP263) Assay detekterar PD-L1-protein i FFPE NSCLC-vävnad färgad med OptiView DAB IHC Detection Kit på ett
BenchMark ULTRA-instrument. VENTANA PD-L1 (SP263) Assay detekterar PD-L1-protein i FFPE NSCLC-vävnad som färgats på
BenchMark ULTRA-instrument, men uPath PD-L1 (SP263) bildanalys för NSCLC-algoritmen validerades med OptiView DAB IHC
Detection Kit som färgades på BenchMark ULTRA-instrumentet.
Image (Bildtagning)
VENTANA DP 200-bildskannern krävs för att skanna bilderna. Bilder måste skannas med 20x förstoring. Om stora delar av bilden är
ur fokus rekommenderas det att objektglasen skannas in igen. Ytterligare information om skanning finns i användarhandboken till
VENTANA DP 200-skannern (art. nr. 1017149SV).
Allmän navigering: uPath enterprise software
uPath enterprise software är avsedd att anpassas efter individuella behov och lokala behov inklusive, men inte begränsat till:
rapportkonfiguration och användargränssnitt. Denna algoritmguide fokuserar på de verktyg som krävs för att använda uPath PD-L1
(SP263) bildanalys för NSCLC-algoritmen. Ytterligare information om uPath enterprise software finns i användarhandboken för uPath
enterprise software.
8 uPath PD-L1 (SP263) bildanalys för NSCLC (icke-småcellig lungcancer) algoritmguide

Patologarbetsflöde
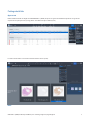
Öppna ett fall
Bilder av NSCLC-vävnad som färgats med VENTANA PD-L1 (SP263) Assay kan nås genom att dubbelklicka på ett fall eller genom att
välja ett fall och trycka på viewer (visning)-fliken inom uPath enterprise software (bild 1).
En skärm med alla bilder associerade med ett fall kommer att visas (bild 2).
Bild2
Bild 1
uPath PD-L1 (SP263) bildanalys för NSCLC (icke-småcellig lungcancer) algoritmguide 9

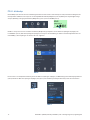
När ett objektglas som är färgat med VENTANA PD-L1 (SP263) Assay har skannats på en VENTANA DP 200-bildskanner vid 20x,
importeras bilden till uPath enterprise software och associeras med ett fall. uPath PD-L1 (SP263) bildanalys för NSCLC-algoritmen
utlöser automatiskt WSA. Tider då WSA-förberäkningssteget ska slutföras beror på serverspecifikationer, bildstorlekar och antal bilder
i kön. När ”waiting to start auto-analysis” (väntar på att starta automatisk analys) visas anger det att bilderna står i kö och ännu inte har
analyserats och ”analyzing” (analyserar) används när WSA utförs (bild 3 och 4). När bilden har analyserats fullständigt via WSA i uPath
enterprise software, kommer ”analysis successful” (analys slutförd) att visas under objektglaset inom vyn i uPath enterprise software
(bild 5). Bilder kan inte göras innan WSA har slutförts.
Rita ROI(er) för hel tumör: Välja tumörområde
Använd verktygsknappen Freehand (Frihand) i rullgardinsmenyn för ROI (bild 6) för att välja tumörområde(n) på den IHC-bildskärmbild
som ska analyseras. Bild 7 illustrerar en bild som har en enda ROI ritad. Ytterligare ROI:er kan ritas. Varje område som väljs visar en ROI i
Slide Panel (Bildpanelen) (bild 8).
Bild 6 Bild 7
Bild 8
Bild 5
Bild4
Bild3
10 uPath PD-L1 (SP263) bildanalys för NSCLC (icke-småcellig lungcancer) algoritmguide

Rita ROI(er) för hel tumör: Exkluderingsområde
När ROI:er ritas kan vissa områden exkluderas. Specifika områden som ska undvikas eller utelämnas samt exempel behandlas i avsnittet
om färgningsegenskaper nedan. Använd exkluderingsverktyget Freehand (Frihand) i rullgardinsmenyn Exclusion (Exkludering) (bild 9)
för att exkludera specifika områden (bild 10). Om stora delar av bilden är suddiga eller ur fokus ska du skanna objektglaset igen.
Exkluderade områden analyseras inte med algoritmen, och de färgade och ofärgade TC i detta område exkluderas från det totala
analysområdet. Om ROI:n redan har analyserats och en exkludering används, måste ROI:n analyseras igen och överläggningen samt
poängen uppdateras därefter.
Det kan vara mycket tidskrävande att rita ett stort antal exkluderingar, och särskilt komplicerade exkluderingar med Freehand (Frihand),
och kan påverka effektiviteten i arbetsflödet samtidigt som det endast har en marginell påverkan på slutpoängen. Om ett fall kräver ett
stort antal exkluderingar ska patologen göra följande:
• Rita flera ROI:er och exkludera delar av vävnad som de ej anser kan poängsättas med minimal användning av Exclusion
(exkludering)-verktyget.
• Begränsa exkluderingar och åsidosätta poängen manuellt med en annan poäng.
Rita ROI(er) för hel tumör: Radering
Om ett valt ROI för hel tumör inte är optimalt kan det raderas. Välj ROI för hel tumör genom att klicka på mitten av ROI:n på bilden
och sedan klicka på knappen Delete (Radera) i Slide Panel (Bildpanelen) (bild 11) eller i objektglaset nära ROI (bild 12). Ett
bekräftelsefönster visas. Välj Confirm (Bekräfta) för att radera vald ROI. Välj Cancel (Avbryt) för att behålla ROI.
Bild 10Bild 9
Bild 11 Bild 12
uPath PD-L1 (SP263) bildanalys för NSCLC (icke-småcellig lungcancer) algoritmguide 11

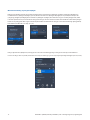
När alla ROI(er) för hel tumör och/eller exkluderingsområden har ritats är bilden redo att analyseras. Välj hela tumörens ROI genom att
klicka på mitten av ROI som ska analyseras eller klicka på ROI i Slide Panel (Bildpanelen). För varje ROI trycker du på knappen Image
Analysis (Bildanalys) antingen på Slide Panel (Bildpanelen) (bild 13) eller bredvid ROI (bild 14).
När PD-L1-analysen är klar visas resultaten i Slide Panel (Bildpanelen) på två platser: under Slide Score (Poäng för objektglas) och
bredvid ROI:er (bild 15). Slide Score (Poäng för objektglas) baseras på en sammanfattning av statusen för tumörcellpositivitet över alla
utvalda ROI(er), vilket är poängen som kommer att visas i rapporten.
PD-L1-bildanalys
Bild 15
Du kan också se mer detaljerad information i panelen för Slide Score (Poäng för objektglas) och ROI-detaljer genom att klicka på panelikonen
(bild 16). Panelen för Slide Score (Poäng för objektglas) visas (bild 17). Om du klickar på samma panelikon döljs också denna information.
Bild 17
Bild 16
Bild 13 Bild 14
12 uPath PD-L1 (SP263) bildanalys för NSCLC (icke-småcellig lungcancer) algoritmguide

PD-L1-bildanalys: Falsk färgöverlagring
När du trycker på knappen Analys av ROI(er) och vävnaden analyserats, kommer en färgöverlagring att visas på ROI:n. I bilden nedan
(bild 18) representerar de röda cirklarna celler som fastställts vara positivt färgade för PD-L1 och de blå cirklar representerar celler som
fastställts vara negativa för PD-L1. När du tar tag i bilden (vänsterklicka med musen och flytta bilden) försvinner överlagringen (bild 19).
När musknappen släpps, visas överlagringen igen (bild 18).
Bild 18
Bild 19
uPath PD-L1 (SP263) bildanalys för NSCLC (icke-småcellig lungcancer) algoritmguide 13

Manuell åsidosättning av poäng för objektglas
Poäng kan åsidosättas manuellt genom att klicka på panelikonen för Slide Score (Poäng för objektglas) i Slide Panel (Bildpanelen)
bredvid Slide Score (Poäng för objektglas) (bild 16). Panelen för Slide Score (Poäng för objektglas) visas (bild 20). Om du väljer edit
(redigering)-knappen (bild 20) på panelen Slide Score (Poäng för objektglas) kan användaren ange en manuell poäng (bild 21). Fältet
Comments (Kommentarer) kan användas för att ange anteckningar om fallet och/eller beslutet att åsidosätta den automatiska poängen.
För PD-L1 kan poäng på 0–100% anges manuellt. Välj ett bekräftelsemeddelande när du anger en manuell åsidosättnings-poäng (bild 22).
Ett bekräftelsemeddelande visas. Välj ”Yes” (Ja).
Poängen på Slide Panel (Bildpanelen) återspeglar den manuella åsidosättningspoängen. Poängen för bildanalys bredvid ROI:erna
kommer inte längre att visas (bild 23). Användaren kan analysera bilden igen genom att trycka på stapeldiagramknappen (bild 13, bild 14).
Bild 21 Bild 22
Bild 20
Bild 23
14 uPath PD-L1 (SP263) bildanalys för NSCLC (icke-småcellig lungcancer) algoritmguide

Se metodbladet för VENTANA PD-L1 (SP263) Assay och tolkningsguiden.
PD-L1-utvärdering
NSCLC neoplastiska celler märkta med VENTANA PD-L1 (SP263) Assay utvärderas med avseende på procent positivitet av TC
med membranfärgning vid vilken intensitet som helst. Den immunohistokemiska färgningen i NSCLC är membranös och/eller
cytoplasmisk och kan uttryckas homogent eller heterogent genom neoplasma. Cytoplasmatisk färgning av tumörceller tas inte med i
TC-positivitetspoängen. Membranfärgning kan ha ett diskontinuerligt, periferiskt eller basolateralt mönster. En isotypmatchad negativ
kontrollantikropp används för att utvärdera närvaron av bakgrund i testprover.
Utvärdering av PD-L1-algoritmfärg
Patologen som använder uPath PD-L1 (SP263) bildanalys för NSCLC-algoritmen ska vara bekant med manuell poängsättning av
VENTANA PD-L1 (SP263) Assay. Patologen ska använda freehand (frihand)-verktyget för att ringa in hela tumörområdet. Patologen
ska hänvisa till den tillhörande H&E och det negativa kontrollglaset innan du använder uPath PD-L1 (SP263) bildanalys för NSCLC-
algoritmen. När du väljer områden som ska analyseras ska du ta hänsyn till de begränsningar som beskrivs i begränsningarna och
områdena för att undvika sektioner. Om patologen inte håller med poängen som tillhandahålls av uPath PD-L1 (SP263) bildanalys för
NSCLC-algoritmen, ska patologen manuellt åsidosätta poängen.
Ej utvärderbara fall inkluderar, men är inte begränsade till, fall med otillräcklig livskraftig tumör, oacceptabel morfologi och störande
bakgrund. NSCLC-fall med tillräckligt livskraftiga TC (som fastställs av den poängsättande läkaren) och ingen störande bakgrund på
PD-L1 IHC-bilden är acceptabla för utvärdering.
Färgningsegenskaper
uPath PD-L1 (SP263) bildanalys för NSCLC (icke-småcellig lungcancer) algoritmguide 15

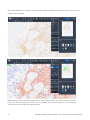
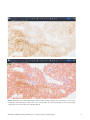
Bild 24: Skannade bilder av IHC-färgade NSCLC-vävnad i uPath enterprise software. Före analys (ovan) och efter analys
(nedan). Den röda överlagringen representerar celler som fastställts att vara positivt färgade TC och blå överlagring
representerar celler som fastställts vara negativt färgade TC.
Bilder av olika färgningsmönster och PD-L1-uttrycksnivåer tillhandahålls i de efterföljande bilderna (24–25). PD-L1-uttryck i TC som
rapporteras i procent (0–100).
16 uPath PD-L1 (SP263) bildanalys för NSCLC (icke-småcellig lungcancer) algoritmguide
Sidan laddas...
Sidan laddas...
Sidan laddas...
Sidan laddas...
Sidan laddas...
Sidan laddas...
Sidan laddas...
Sidan laddas...
Sidan laddas...
Sidan laddas...
Sidan laddas...
Sidan laddas...
Sidan laddas...
Sidan laddas...
Sidan laddas...
Sidan laddas...
Sidan laddas...
Sidan laddas...
-
 1
1
-
 2
2
-
 3
3
-
 4
4
-
 5
5
-
 6
6
-
 7
7
-
 8
8
-
 9
9
-
 10
10
-
 11
11
-
 12
12
-
 13
13
-
 14
14
-
 15
15
-
 16
16
-
 17
17
-
 18
18
-
 19
19
-
 20
20
-
 21
21
-
 22
22
-
 23
23
-
 24
24
-
 25
25
-
 26
26
-
 27
27
-
 28
28
-
 29
29
-
 30
30
-
 31
31
-
 32
32
-
 33
33
-
 34
34
-
 35
35
-
 36
36
-
 37
37
-
 38
38
Roche uPath PD-L1 image analysis for NSCLC IVD Algorithm Användarmanual
- Typ
- Användarmanual
på andra språk
Relaterade papper
-
Roche uPath IVD Användarmanual
-
Roche BenchMark ULTRA Användarmanual
-
Roche uPath IVD Användarmanual
-
Roche BenchMark XT/LT Användarmanual
-
Roche BenchMark ULTRA Interpretation Guide
-
Roche BenchMark GX Användarmanual
-
Roche BenchMark ULTRA Användarmanual
-
Roche iScan HT RUO Användarmanual
-
Roche Virtuoso Användarmanual
-
Roche Virtuoso Användarmanual






































